📖 DataFrames
I nostri metadati come DataFrame
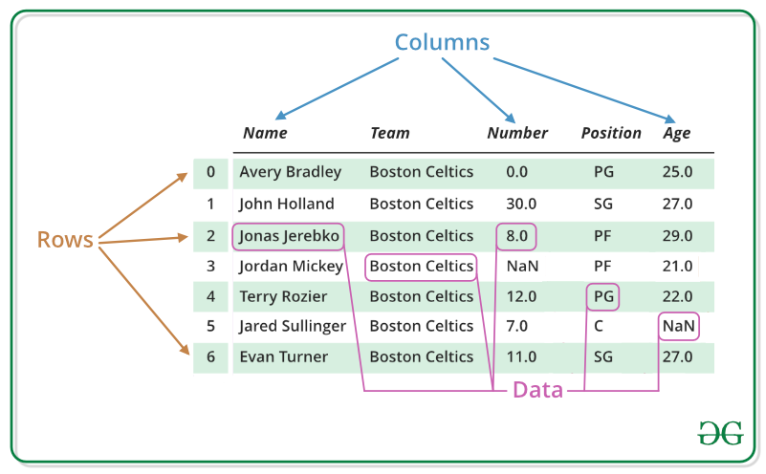
Un DataFrame rappresenta una tabella bidimensionale costituita da righe e colonne, simile a un foglio di calcolo Excel. Il DataFrame è uno dei vari tipi di variabile che potrai trovare in Python! In questo workshop, il DataFrame costituisce la variabile principale: infatti nei progetti che affronteremo, i metadati da curare saranno trattati come DataFrame. Quindi, gran parte del nostro lavoro consisterà nella manipolazione e nell’analisi dei metadati strutturati in DataFrame.
Importare il file di metadati come DataFrame
Per prima cosa dobbiamo leggere il file di metadati come un DataFrame:
import pandas as pd
df = pd.read_csv("ERP020892_df.tsv", sep="\t")
print(df)
study_accession secondary_study_accession sample_accession \
0 PRJEB18925 ERP020892 SAMEA46804918
1 PRJEB18925 ERP020892 SAMEA46799668
2 PRJEB18925 ERP020892 SAMEA46802668
3 PRJEB18925 ERP020892 SAMEA46803418
4 PRJEB18925 ERP020892 SAMEA46798918
5 PRJEB18925 ERP020892 SAMEA46801168
secondary_sample_accession experiment_accession run_accession \
0 ERS1500521 ERX1845438 ERR1781715
1 ERS1500514 ERX1845431 ERR1781708
2 ERS1500518 ERX1845435 ERR1781712
3 ERS1500519 ERX1845436 ERR1781713
4 ERS1500513 ERX1845430 ERR1781707
5 ERS1500516 ERX1845433 ERR1781710
submission_accession tax_id scientific_name_exp instrument_platform \
0 ERA787503 539655.0 human skin metagenome ILLUMINA
1 ERA787503 539655.0 human skin metagenome ILLUMINA
2 ERA787503 539655.0 human skin metagenome ILLUMINA
3 ERA787503 539655.0 human skin metagenome ILLUMINA
4 ERA787503 539655.0 human skin metagenome ILLUMINA
5 ERA787503 539655.0 human skin metagenome ILLUMINA
... barcode_y center_name linker primer run_prefix \
0 ... ACTCAGATACTC CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
1 ... AGCACGAGCCTA CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
2 ... AGACGTGCACTG CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
3 ... ACGGATCGTCAG CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
4 ... AACTCGTCGATG CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
5 ... AGCTGACTAGTC CCME GT GTGCCAGCMGCCGCGGTAA s_6_1_sequences
collection_timestamp_y dna_extracted_y physical_specimen_location_y \
0 04/06/2010 True UCSDMI
1 04/06/2010 True UCSDMI
2 04/06/2010 True UCSDMI
3 04/06/2010 True UCSDMI
4 04/06/2010 True UCSDMI
5 04/06/2010 True UCSDMI
physical_specimen_remaining_y sample_location
0 False UCSDMI
1 False UCSDMI
2 False UCSDMI
3 False UCSDMI
4 False UCSDMI
5 False UCSDMI
[6 rows x 193 columns]
Ecco, abbiamo iniziato importando la libreria Pandas per poter sfruttare le sue funzioni. La prima funzione che abbiamo esplorato è read_csv, essenziale per leggere un file tabulare (csv, tsv, ecc.) e convertirlo in un DataFrame. In questa funzione, abbiamo indicato il nome del file tra virgolette (perché è una stringa, un altro tipo di variabile in Python!) e il separatore del file. Nel nostro caso, i metadati sono in formato TSV (Tab-Separated Values), cioè i dati sono separati da un tab. Per questo motivo, nella funzione dobbiamo aggiungere dopo il nome del file, l’opzione sep="\t" per indicare alla funzione che i dati sono separati da tabulazione.
Ah, nota che abbiamo usato una funzione predefinita di Python: print(). Questa funzione è utilizzata per visualizzare ciò che viene inserito tra le sue parentesi. In questa caso gli abbiamo chiesto di mostrarci il DataFrame df.